姜卫红研究组在放线菌天然产物的合成生物学使能技术方面取得新进展
2017年1月11日,国际学术期刊《Metabolic Engineering》杂志在线发表了中科院上海植物生理生态研究所姜卫红研究组题为“Multiplexed site-specific genome engineering for overproducing bioactive secondary metabolites in actinomycetes”的研究论文。该工作成功地发展了通用型的放线菌天然产物生物合成基因簇的多拷贝稳定扩增与高效编辑新方法,为微生物药物高产菌株的分子育种以及新化合物的挖掘提供了新的思路和策略。
微生物天然产物种类繁多,据统计至今总计有18000多种被鉴定,包括广泛应用的抗生素、抗肿瘤药物、免疫抑制剂、杀虫剂等,其中50%以上由放线菌产生。因此,放线菌来源的天然产物在医药工业中占据重要的地位。目前,放线菌天然产物高产菌株的选育还是主要依赖于传统的理化诱变筛选,费时费力,发展高效、先进的菌株分子选育技术十分必要。
由始旋链霉菌(Streptomyces pristinaespiralis)产生的普那霉素(Pristinamycin)是一种抗耐药菌抗生素,包含普纳霉素I(PI)和 II(PII)两个组分,其衍生物已被批准用于治疗多种临床耐药性病原菌,如耐甲氧西林葡萄球菌与耐万古霉素肠球菌等。在前期研究工作中,姜卫红研究组的博士研究生李雷和芦银华研究员通过组合代谢工程与发酵工艺优化,初步提高了普那霉素II(PII)的合成能力。本研究中,他们进一步创新技术,建立了基于“一个整合酶-多个attB位点”理念的放线菌天然产物生物合成基因簇多拷贝整合的新方法MSGE。运用该方法,实现了PII生物合成基因簇的高效、快速扩增,获得了多达5个PII生物合成基因簇拷贝的工程菌SBJ1005。该工程菌的最高产量达到2.2 g/L,与出发菌株相比,提高了11倍,已基本接近产业化要求。在此过程中,他们还组合运用体外CRISPR/Cas9系统与Gibson等温一步法,开发了一种新型的基因簇编辑技术CGE,为放线菌天然产物生物合成体系的优化提供了重要的技术手段。
此外,基于MSGE理念,他们还构建了一系列含有多个外源ΦC31 attB位点的天蓝色链霉菌异源表达底盘宿主,实现了氯霉素或抗癌小分子YM-216391合成基因簇多达4个拷贝的高效整合。与现有放线菌宿主相比,运用新底盘细胞测试的2种天然产物的产量提升了2-23倍。这些优秀底盘细胞的开发对于通过Genome mining策略发现新型天然产物的研究工作具有重要意义。
该工作得到了国家科技部973与863项目的资助。
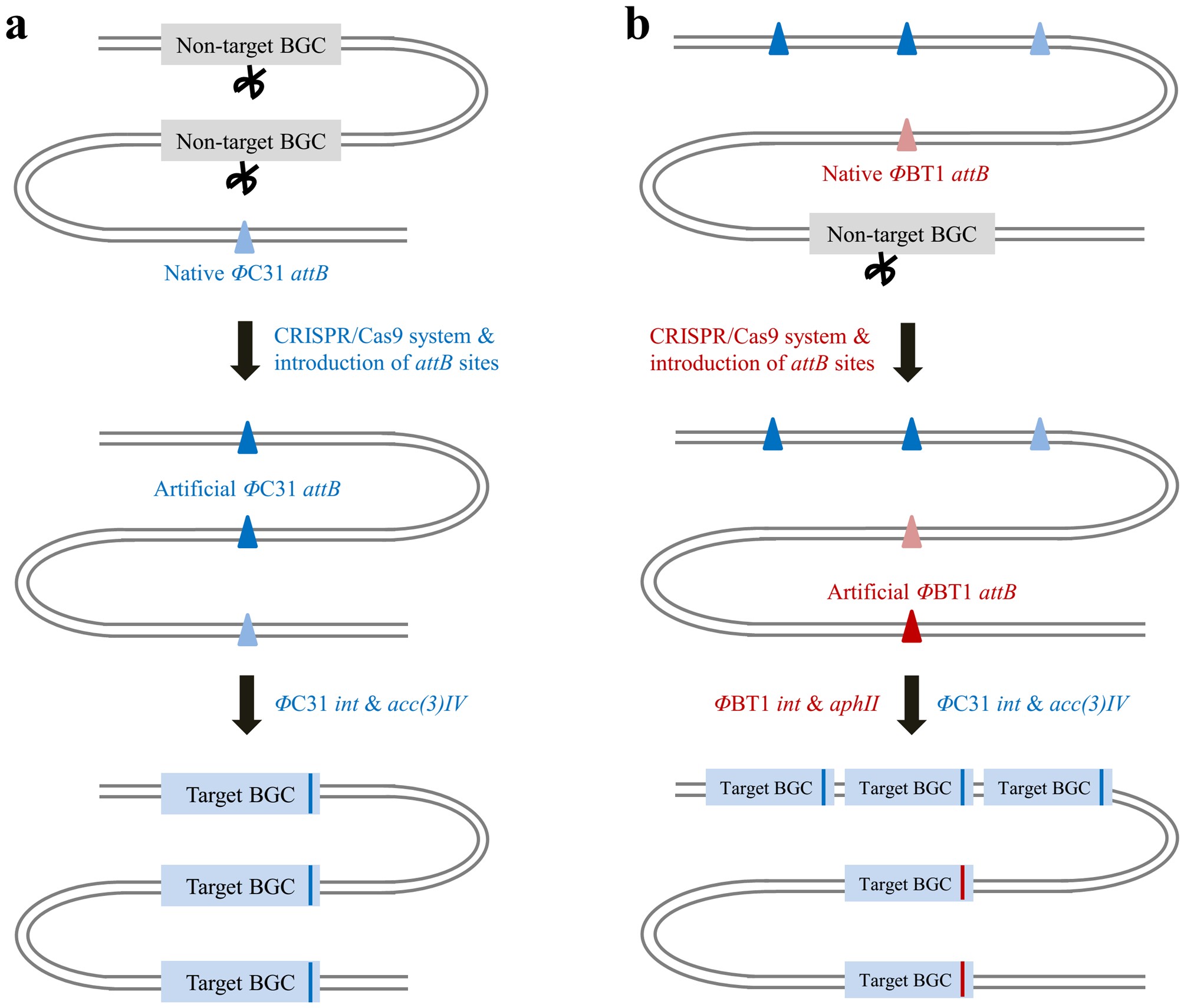
图示:放线菌天然产物生物合成基因簇多拷贝稳定整合的新方法(MSGE)