姜卫红研究组揭示革兰氏阳性细菌CcpA蛋白的调控新机制
1月24日,国际学术期刊《mBio》在线发表了我所姜卫红研究组题为“A flexible binding site architecture provides new insights into CcpA global regulation in Gram-positive bacteria”的研究论文。该工作揭示了革兰氏阳性细菌全局调控蛋白CcpA的一种新型、高度可变的结合靶基序cre-var及其相关分子调控机制,为认识和理解其调控的多样化和精细化提供了新视角。
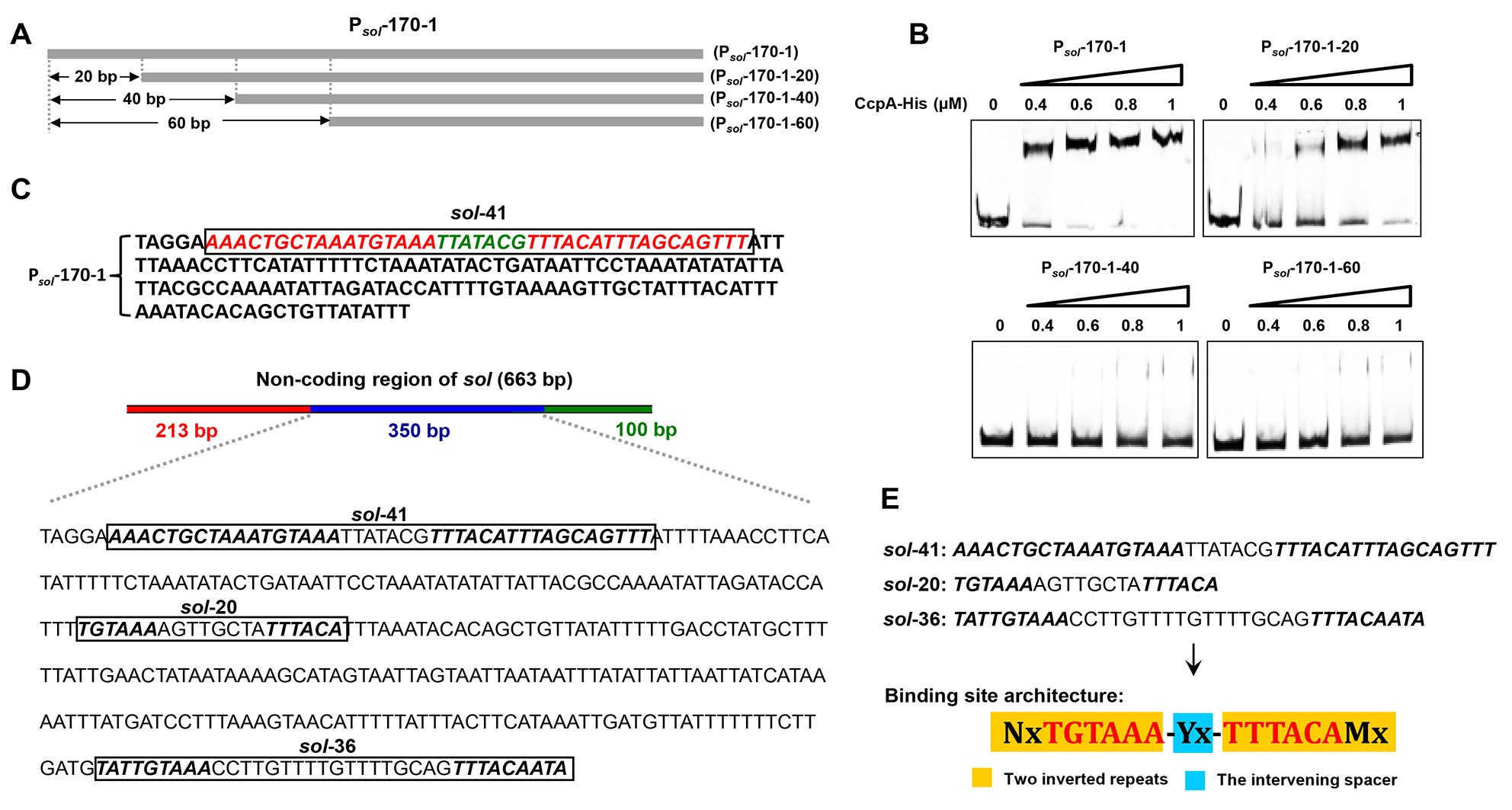
CcpA是革兰氏阳性细菌中重要的全局性转录调控因子,参与控制细胞的多项生理过程。它通过识别并结合至靶基因的特定位点执行其转录调控功能,进而形成复杂的调控网络,这类结合位点被称为cre(catabolite response elements)序列。目前已发现的被CcpA识别的cre通常是14-16个核苷酸的反向回文序列,其基序在长度和核苷酸组成上均比较相近。
课题组前期以革兰氏阳性菌—丙酮丁醇梭菌为研究对象,通过比较转录谱分析发现,大量受CcpA影响的基因在非编码区和编码区均不存在典型的cre位点,由此引出的问题是:这些基因中是否存在受CcpA直接调控的新位点?如果有,这些调控位点的结构与功能如何?为此,博士生杨云鹏在姜卫红和顾阳两位研究员的指导下,以其中一个转录受CcpA显著影响的sol操纵子为突破口进行深入探究,发现了一种非典型的、具有特殊结构的结合基序。该基序与典型的cre差异较大,呈现高度可变性,即除了两端各6个核苷酸的保守反向回文序列(TGTAAA/TTTACA)外,其中间间隔区和向外延伸部分在长度和核苷酸组成上均是可变的,该新型结合基序被命名为cre-var。进一步的研究表明,cre-var两端的保守反向回文序列和中央间隔区的核苷酸及长度变化会显著影响其与CcpA的亲和力,从而揭示了其“柔性结构”的功能。随后的全基因组搜索发现,丙酮丁醇梭菌中存在一百多个cre-var位点,它们既可以单独行使功能也可以与经典的cre协同发挥作用,使得CcpA的调控更全面、灵活和精细。尤其重要的是,cre-var在其它革兰氏阳性菌中也大量存在,包括致病型梭菌、芽孢杆菌、链球菌、肠球菌和乳酸菌等,具有普适性,也为在这些细菌中开展相关研究奠定了基础。
本研究得到国家基金委、国家高技术研究发展计划(863计划)的资助。