赵国屏研究组在Cpf1蛋白切割机理方面取得新进展
2017年1月11日,国际学术期刊《Nucleic Acids Research》杂志在线发表了中科院上海植物生理生态研究所赵国屏研究组题为“The CCTL (Cpf1-assisted Cutting and Taq DNA ligase-assisted Ligation) method for efficient editing of large DNA constructs in vitro”的研究论文。该工作鉴定了Cpf1蛋白的精确切割位点,并基于该切割特性开发新的DNA无缝拼接方法。
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)是存在于原核生物中的获得性免疫系统,CRISPR相关蛋白Cas9已广泛应用于基因组编辑和许多其他应用当中。Cpf1蛋白隶属于II类 type V CRISPR系统。相比于Cas9蛋白,Cpf1蛋白具有类似的基因组编辑效率,但具有较低的脱靶效应,在基因治疗应用中具有巨大的潜力。不同于Cas9,Cpf1由单个crRNA指导,并利用富含T的protospacer相邻基序(PAM)序列切割双链DNA(dsDNA)靶标,形成5-nt粘性末端。Cpf1的切割特性使得其成为有效的体外DNA拼接工具,并且最近的研究中报道了基于Cpf1消化和T4DNA连接酶介导的连接建立的DNA拼接标准C-Brick。
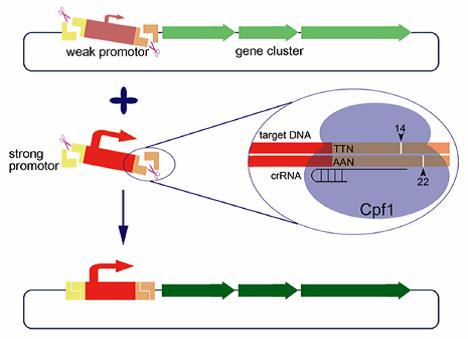
在本研究中,研究人员发现,Cpf1切割靶标DNA并不像之前报道的那样,只切割靶标DNA互补链的23位和非互补链的18位,而是在非互补链的14位到18位形成多个切割位点。除此之外,本文研究人员还发现Cpf1的切割位点受到crRNA spacer序列长度的影响。当spacer序列长度>=20的时候,Cpf1倾向于切割非互补链的18位,而当spacer序列长度<20的时候,Cpf1倾向于切割非互补链的14位。基于Cpf1在较短spacer长度的crRNA介导下,可以特异切割靶标DNA的14位与22位,形成8-nt的长黏性末端的特性,结合Taq DNA连接酶高精度的连接特性,研究人员将其开发为一个大DNA片段体外无缝编辑的新工具。在应用实例中,研究人员成功将30kb的放线紫红素合成基因簇内部调控基因actII-orf4的启动子进行了原位的替换。经过反应条件的优化,替换的阳性率达到70%以上。该方法为大片段体外编辑提供了一个高效工具。