逆境中心杜嘉木研究组与遗传发育所曹晓风研究组合作揭示KDM5亚家族组蛋白去甲基化酶的底物识别机制
12月12日,The Plant Cell杂志在线发表了中科院植物逆境生物学研究中心杜嘉木课题组和中科院遗传发育所曹晓风院士课题组合作完成的题为“Structure of the Arabidopsis JMJ14-H3K4me3 Complex Provides Insight into the Substrate Specificity of KDM5 Subfamily Histone Demethylases”的研究论文,该研究利用结构生物学,生物化学和细胞生物学等手段揭示了KDM5亚家族组蛋白去甲基化酶的底物识别机制。
组蛋白修饰在生物体生长发育过程中发挥着至关重要的作用,它与基因沉默,基因激活,肿瘤生成,染色体形态建成以及遗传物质修复等生物学问题密切相关。对其分子机制的研究为我们探索生命本质以及开发相应的靶点药物提供了理论依据。
先前的研究表明拟南芥JMJ14是一个含有jumonji结构域的组蛋白H3K4me3的去甲基化酶,从序列分析上属于KDM5亚家族。人源的KDM5亚家族基因可以促进肿瘤生成及产生耐药性,因而是重要的抗癌药物靶标,但是不管是植物还是动物中KDM5亚家族去甲基化酶的其底物识别机制一直不是很清楚。拟南芥的JMJ14已报道具有调控开花、RNA介导的基因沉默以及DNA甲基化等重要的生理过程,但是其底物识别机制也不是很清楚。
在该研究中,研究人员使用了更为精准的酶活测定系统,质谱结果表明JMJ14是一个H3K4me3和me2的去甲基化酶,而对me1作用不明显。而后利用X射线晶体衍射的方法解析了JMJ14处于apo状态和底物复合物状态的结构。JMJ14主要有jumonji、helical以及C5HC2三个结构域组成,其中jumonji以及C5HC2结构域上的3个酸性氨基酸与组蛋白小肽上H3R2和H3Q5发生了大量的氢键以及盐键的相互作用,形成特异性识别。将这几个酸性氨基酸突变之后,JMJ14的体内酶活显著下降,表明这几个氨基酸确实参与了底物识别。此外,由于人源KDM5家族蛋白是一个重要的抗癌药物靶标,而其底物识别机制还不是很清楚。通过氨基酸序列和三维结构比对发现,JMJ14参与底物识别的氨基酸在KDM5B中也非常保守,暗示这它们采用了相似的底物识别机制。之后将KDM5B中相应的氨基酸突变后其酶活明显下降,证明了以上猜想。该项工作不仅解析了以JMJ14为代表的KDM5亚家族H3K4me3去甲基化酶的去甲基化机制,还为以人源KDM5为靶标的抗癌药物开发提供了理论基础。
该论文第一作者是逆境中心博士生杨振林,逆境中心杜嘉木研究员和遗传发育所曹晓风院士是该论文的共同通讯作者。普渡大学的陶纬国教授,中科院上海生化细胞所的陈德桂研究员及南方科技大学的李思思副教授为该论文提供了重要的支持和帮助。遗传发育所邱琦博士和逆境中心陈小梅博士等参与了课题的相关部分。逆境中心质谱平台为本研究提供了酶活实验帮助。上海同步辐射光源国家蛋白质设施BL19U1(SSRF)为数据收集提供了及时有效的支持。该研究工作得到了科技部国家重点研发计划,国家自然科学基金委,以及中科院相关经费的大力资助。
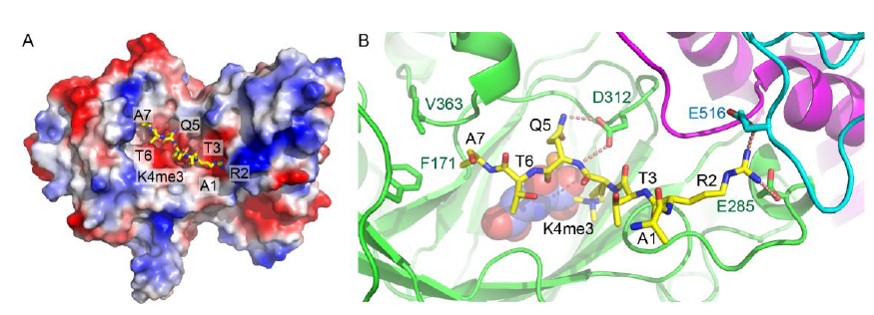
JMJ14特异识别并催化组蛋白H3K4me3的分子机制
A.JMJ14催化结构域的表面电荷分布,组蛋白H3K4me3以棒状模型展示。
B.JMJ14特异识别组蛋白第二位的精氨酸,第五位的谷氨酰胺和第7位的丙氨酸。