肖友利研究组通过发展分子探针技术实现植物天然产物合成途径解析的新策略
植物天然产物经过长期的分子进化,以其多样独特的分子结构,在生命活动中扮演着重要的角色,不仅在其宿主内源中发挥着信号传导、营养、抗逆和防御等生理作用,而且在异源也具有各种药理活性,是药物研发的重要来源。但是一些具有生物活性的植物次生代谢产物(特殊营养成分)往往在植物中含量极少;而且生物合成途径的缺失,极大阻碍了这些重要活性化合物通过现代合成生物学的方法进行规模化的生物制造。不同于原核天然产物合成基因的成簇形式,植物天然产物合成途径相关的基因元件相对离散性的分布在植物染色体上,极大地限制了利用基因敲除和突变的传统策略进行植物天然产物合同途径解析的效率。而随着高通量新一代测序技术的高速发展,植物基因组和转录组数据得到快速富集,远远超出天然产物合成途径相关功能基因组的解析速度。
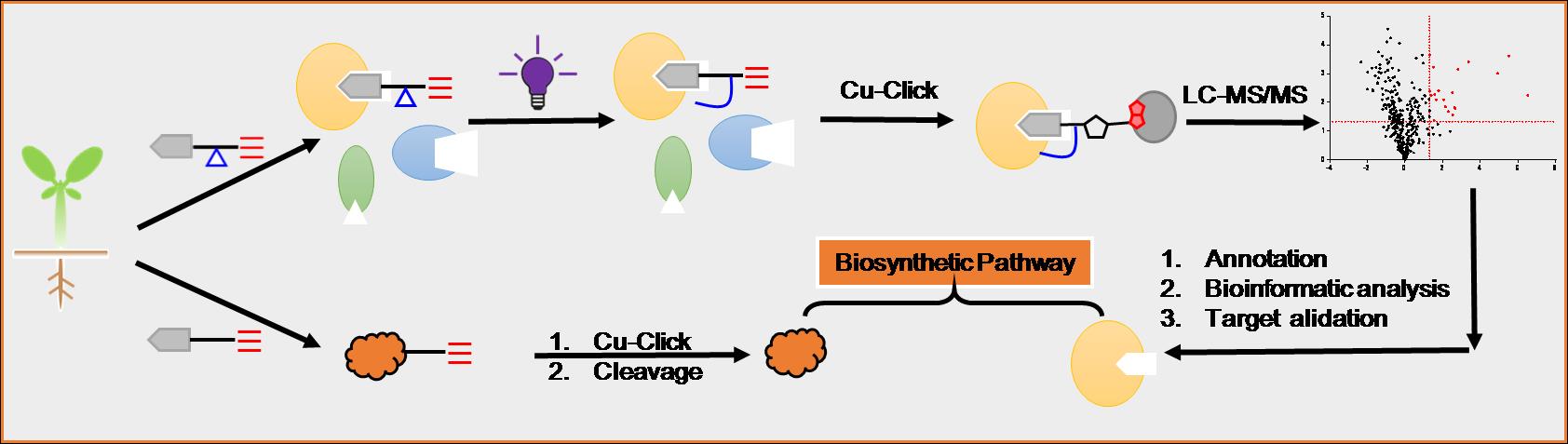
针对这一不对等的数据信息积累和发展,为获得植物天然产物的合成途径高效解析和新元件挖掘,肖友利研究组致力于发展功能分子探针进行合成途径相关蛋白酶和代谢物解析的新策略。作为一个案例初探,该研究组李伟超博士、周怡青副研究员、尤文静和杨盟权同学等首先利用活性分子探针导向的定量化学蛋白质组学策略,以二萜贝壳杉稀骨架衍生的甜菊醇(steviol)为基本结构设计化学小分子探针,分别实现在非模式植物的富产甜菊糖苷同源宿主(甜叶菊)和不产该类化合物异源(拟南芥)模式植物的活性蛋白质组中简单快捷的标记目标作用蛋白,从与甜菊醇骨架结合的蛋白中筛选具有特定催化活性的糖基转移酶。同时他们还基于非模式植物的转录组数据构建了蛋白质组数据库,从而依托成熟的质谱技术解析蛋白质序列,高通量获取甜菊糖苷生物合成途径中的多种转糖酶元件。该研究发现,甜叶菊来源的UGT73E1、UGT76G3和拟南芥来源的UGT73C1等糖基转移酶可以作为潜在的生物合成元件获得新颖的代谢产物。进一步利用光亲和探针标记技术,他们还成功解析了各转糖酶的底物结合位点,结合分子对接模拟计算对二萜化合物的糖基转移酶的底物专一性识别机制进行了解析。同时基于分子探针的设计,通过先活性反应对代谢合成物富集然后解离代谢组分析的策略获得关键代谢相关信息。以上两方面相关功能蛋白酶和代谢物的解析信息实现了合成途径的成功绘制。
综上,肖友利研究组通过以贝壳杉烯骨架糖苷类化合物为模型的植物次生代谢产物糖基化研究,建立了研究植物天然产物生物合成途径解析和元件挖掘的通用模型。以上成果分别于6月4日和12日在线发表于美国化学学会化学生物学 (ACS Chemical Biology) 以及上英国皇家化学学会旗下化学通讯 (Chemical Communications)相关领域权威期刊上,相关方法及结果已提交专利申请。该研究工作得到瑞士联邦理工学院袁曙光博士和中科院分子植物科学卓越创新中心/植物生理生态研究所王勇研究员的合作支持以及自然科学基金,中科院先导B预演项目,国际合作大科学培育计划和上海市科委等经费的资助。小分子核磁和质谱的测试工作得到了中科院分子植物科学卓越创新中心/植物生理生态研究所公共技术服务中心代谢组学与蛋白互作技术平台的支持,分子模拟计算在波兰华沙的数学与计算模型交叉中心完成。作者同时也对山东大学侯丙凯教授和四川农业大学吴卫教授提供的生物材料及生物信息进行了致谢。
论文链接:
1.https://pubs.acs.org/doi/10.1021/acschembio.8b00285
2.http://pubs.rsc.org/en/content/articlelanding/2018/cc/c7cc09951g