植物逆境中心杜嘉木研究组与威斯康辛大学麦迪逊分校钟雪花研究组合作揭示SHL蛋白对于抑制和活性组蛋白修饰的识别机制
6月21日,《Nature Communications》 杂志在线发表了中科院分子植物卓越中心/植物生理生态研究所植物逆境生物学研究中心植物分子遗传国家重点实验室杜嘉木研究组和威斯康辛大学麦迪逊分校钟雪花研究组合作完成的题为“Dual recognition of H3K4me3 and H3K27me3 by a plant histone reader SHL ”的研究论文,该研究综合利用结构生物学、生物化学、植物分子遗传学和基因组学等手段联合揭示了植物特有的组蛋白识别蛋白SHL通过自身BAH结构域和PHD结构域分别识别抑制性组蛋白修饰H3K27me3和活性组蛋白修饰H3K4me3的分子机制。
组蛋白的转录后修饰作为表观遗传中重要的调控机制之一,在包括基因表达调控等多种生物学过程中起着重要作用。组蛋白修饰的识别因子具有对于不同的组蛋白修饰特异性识别的能力,并进而在染色体的状态调控中起到重要作用。通常识别蛋白含有多个组蛋白识别结构域,这些结构域通常以串联的方式存在,并且在多价染色质结合中起作用。
拟南芥SHORT LIFE (SHL)同时具有两个潜在的组蛋白识别因子:BAH 及PHD 结构域,在拟南芥的开花及种子休眠起到重要调控作用。在之前报道中表明SHL可以通过抑制开花过程中整合因子SUPPRESSION OF OVEREXPRESSION OF CO1 (SOC1) 的表达进而抑制开花。尽管SHL蛋白起到了抑制开花的作用,其具体的分子机制还不是很清楚。
本研究中,研究人员首先通过组蛋白肽段微阵列芯片技术发现SHL蛋白可以识别组蛋白激活修饰H3K4me3 及组蛋白抑制修饰H3K27me3,通过ITC 证实SHL可以识别甲基化的H3K4和H3K27。紧接着,研究人员利用X射线晶体衍射的方法分别解析了SHL与H3K4me3及H3K27me3的复合物结构,并结合其他生化实验证实证明了SHL能够分别通过其PHD和BAH域独立识别活性组蛋白标记H3K4me3和抑制标记H3K27me3。通过Chip-seq实验证实SHL在体内分布与共富集H3K4me3和H3K27me3的区域相关。遗传实验证明SHL蛋白中的BAH-H3K27me3和PHD-H3K4me3结合位点突变会导致不能回补早期shl突变体开花表型,表明SHL与H3K27me3和H3K4me3的结合在体内是具有重要功能。
植物逆境中心二年级研究生吕莘辰同学是该轮文共同第一作者,植物逆境中心杜嘉木研究员及威斯康辛大学麦迪逊分校钟雪花博士为共同通讯作者。上海同步辐射光源国家蛋白质设施BL19U1(SSRF)为数据收集提供了及时有效的支持。该研究工作得到了科技部国家重点研发计划,国家自然科学基金委,以及中科院的经费资助。
文章链接:https://www.nature.com/articles/s41467-018-04836-y
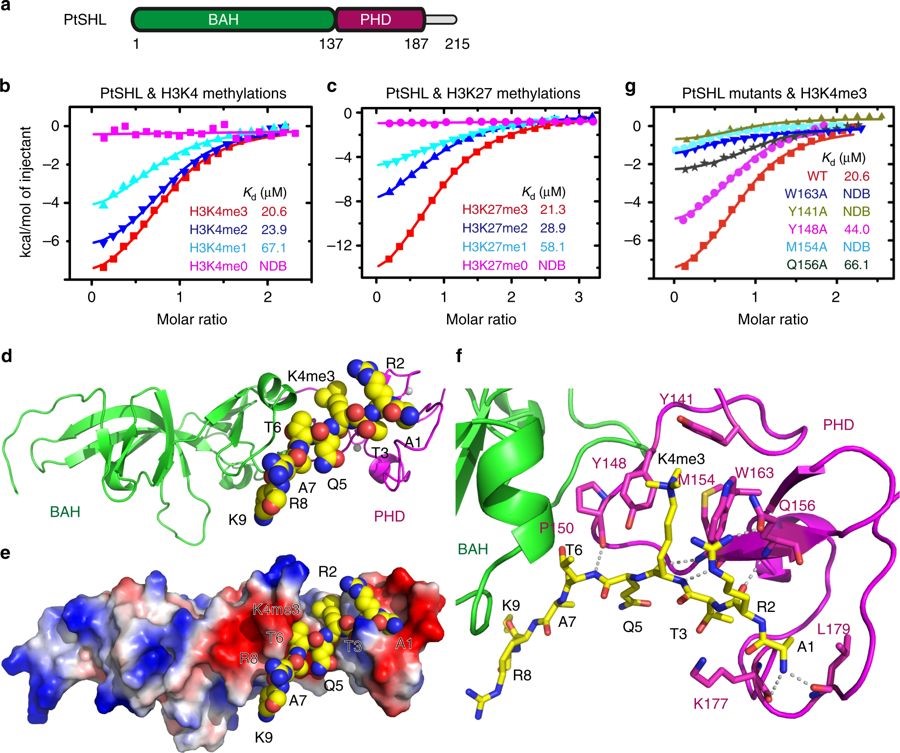
图1.SHL及H3K4me3复合物结构
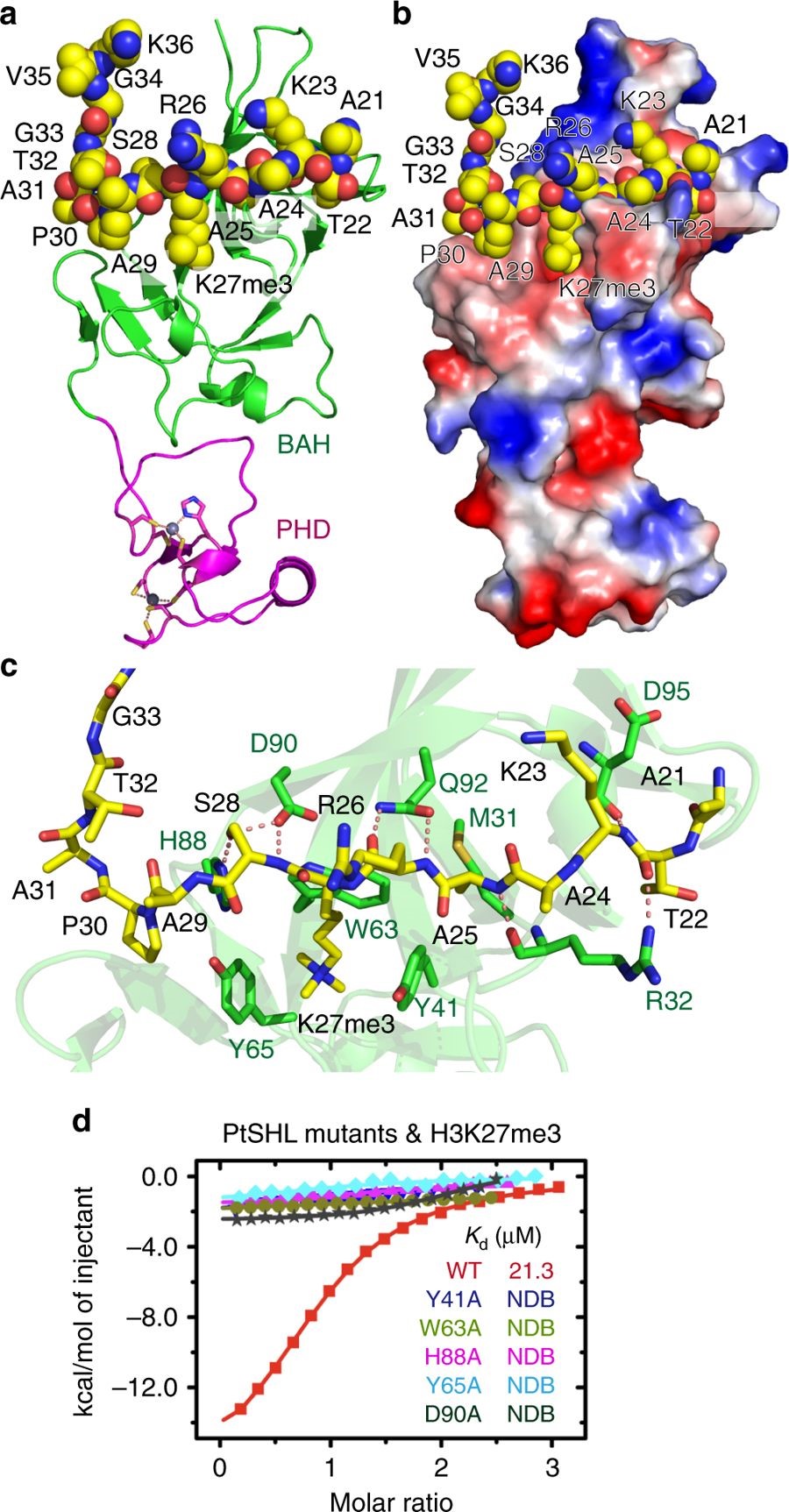
图2.SHL及H3K27me3复合物结构