姜卫红研究组建立放线菌天然产物合成基因簇多位点染色体插入新技术
2018年12月5日,国际学术期刊《Metabolic Engineering》在线发表了中科院分子植物科学卓越创新中心/植物生理生态研究所姜卫红研究组题为“aMSGE: advanced multiplex site-specific genome engineering with orthogonal modular recombinases in actinomycetes”的论文。该研究创建了一种高效、通用的放线菌天然产物合成基因簇多位点染色体插入的新技术,为微生物药物的研究与开发提供了又一种先进的操作工具。
放线菌天然产物具有多元的化学结构与广谱的生物学活性,已被广泛用作杀虫剂、抗肿瘤药物与免疫抑制剂等,为人类健康以及农牧业病虫害防治作出了巨大贡献。然而,放线菌重要药物产生菌通常发酵单位较低且遗传操作困难。目前,高产菌的构建还主要依赖常规诱变筛选,效率低且工作量大,因此亟需发展先进高效的分子育种平台,从而实现其生物制造技术的升级换代。
为优化目标产物的生物合成,目前广泛使用的代谢工程策略包括强化合成途径、删减竞争支路、优化调控网络、反应区室化以及实时动态响应等。不言而喻,通过增加生物合成基因簇的拷贝数以强化目标产物合成是最为直接、有效的策略之一。2017年,姜卫红研究组基于“一个整合酶-多个attB位点”的理念,建立了一种新的放线菌天然产物合成基因簇扩增方法MSGE(Li et al., Metab Eng, 2017)(图1A左)。该方法显著加快了异源表达底盘细胞以及一些易操作工业菌株的构建,然而需多轮引入人工attB位点,且不适用于遗传操作困难的放线菌。本论文中,作者巧妙利用放线菌基因组天然存在的多个attB位点与一系列正交的位点特异性重组系统,基于“多个整合酶-多个attB位点”的理念,开发了一种更为高效、普适的基因簇扩增新方法aMSGE(图1A右)。新方法仅需将多套整合系统直接添加至含有目标基因簇的载体上,即可一步实现基因簇多位点染色体插入。为加快aMSGE方法的实施,还构建了一套“即插即用”的多拷贝整合工具箱(图1B)。最后,利用新建立的aMSGE方法,作者分别在天蓝色链霉菌与冰城链霉菌中,快速实现了丙二酰辅酶A合成基因与5-酮米尔贝霉素(杀虫剂米尔贝肟前体)合成基因簇3个拷贝的染色体整合,其终产物放线紫红素与5-酮米尔贝霉素产量分别提升4.6与1.9倍,体现了该方法的普适性。
作为新一代放线菌合成生物技术,aMSGE方法无需在目标基因(簇)扩增前对染色体进行任何遗传修饰,因此大幅节省分子育种时间,适用于遗传操作困难的工业菌株。由于位点特异重组系统广泛分布于多种细菌中,aMSGE方法也可拓展到其他细菌的基因组工程改造,从而加快化学品、生物燃料及药物的高效合成。
博士后李雷为本论文的第一作者,上海师范大学芦银华研究员为共同通讯作者。该研究获得了重大新药创制科技专项、博士后创新人才支持计划、上海市自然科学基金等项目的资助。
论文链接:https://doi.org/10.1016/j.ymben.2018.12.001
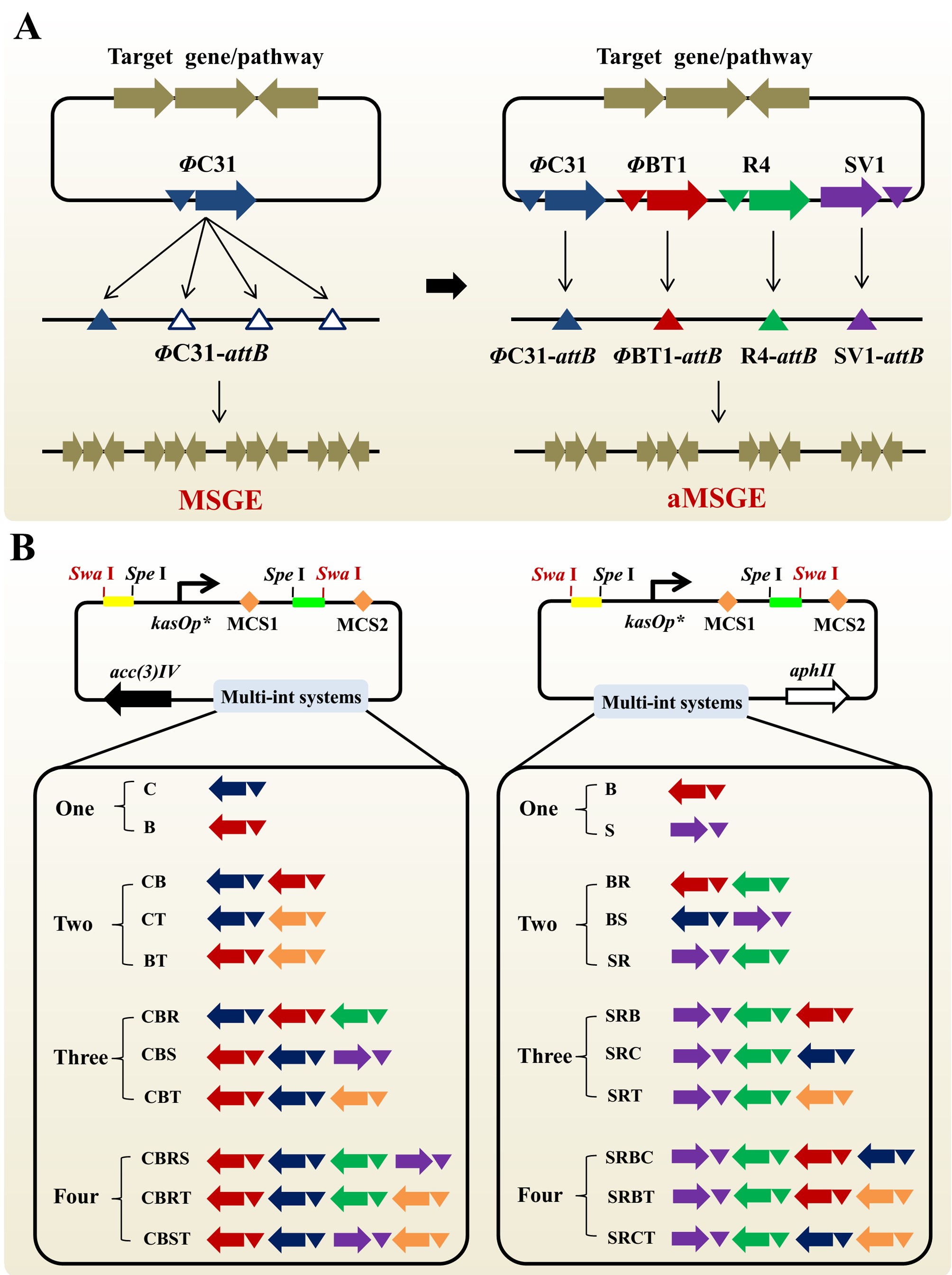
图1 放线菌天然产物合成基因簇多位点染色体插入的新技术