姜卫红研究组建立食气梭菌大片段基因簇染色体整合表达新技术
2019年1月8日,国际学术期刊《Metabolic Engineering》在线发表了中科院分子植物科学卓越创新中心/植物生理生态研究所姜卫红研究组题为“Phage serine integrase-mediated genome engineering for efficient expression of chemical biosynthetic pathway in gas-fermenting Clostridium ljungdahlii”的论文。该研究在化能自养型一碳气体利用菌——食气梭菌中建立了一种基于噬菌体位点特异性重组机制的大片段基因簇染色体整合表达的新技术,为新型一碳气体化能细胞工厂的创建和研究提供了有用的遗传操作工具。
一碳气体(如CO2、CO)是储量巨大的游离性碳资源。近年来,为了拓展它们的利用途径并大力发展绿色生物制造,通过生物转化一碳气体以合成各类化学品和能源物质的技术路线受到广泛关注。迄今已知的可利用一碳气体的微生物按能量需求主要可分为化学能、光能以及电能自养等类型。食气梭菌是一类主要的化能自养菌,以氢气或其他还原性化合物作为能量供体,其气体发酵过程与传统工业发酵设备的匹配性较好,具有良好的应用前景。然而,食气梭菌的天然产物种类少,发酵单位较低且遗传操作困难。因此,亟待发展高效的分子技术平台,用于其代谢工程设计、改造以及合成生物学研究,进而创建更为高效和产物多样化的人工固碳体系,实现一碳气体生物转化利用能力的提升。
为构建高效且产物多样化的食气梭菌细胞工厂,无论是天然产物的强化合成,还是异源途径的适配表达,大片段代谢途径基因簇在染色体上的高效整合与编辑都是必须用到的有效策略,其遗传稳定性显著优于借助游离性质粒载体的方法。2016年,姜卫红研究组率先在食气梭菌Clostridium ljungdahlii中建立了CRISPR-Cas9介导的基因组编辑技术(Huang et al., ACS Synth Biol, 2016)。该技术可用于食气梭菌染色体上基因的删除或小片段DNA在染色体上的整合,但受限于较低的同源重组效率,并不能实现在染色体上通过重组机制引入大片段目标产物合成基因簇的目的。为解决上述难题,本研究首先借助于CRISPR-Cas9基因编辑系统,在食气梭菌染色体上整合了来自于两套异源噬菌体整合系统的attB位点。在此基础上,通过整合酶介导的attP-attB重组机制实现了外源基因簇在染色体上任一attB位点的高效插入(100%),从而实现新产物的合成。此外,新方法还通过基于“双整合酶-双attP/attB”策略的两次重组以及CRISPR-Cas9定向切割的筛选作用,实现了特定目标基因簇在染色体上的整合,解决了使用单套attP/attB系统时引入其他“杂质DNA”的缺陷,从而确保基因簇的稳定表达。最终,作者在食气梭菌Clostridium ljungdahlii的染色体上成功整合了来源于丙酮丁醇梭菌的丁酸合成基因簇。在单批气体发酵中,工程菌的丁酸产量达到1.01 g/L, 其产物浓度和合成速率均高于已报道的食气梭菌,且经过10余代次的连续培养仍能保持这一发酵水平,显示了良好的遗传稳定性。
上述研究结果弥补了现有食气梭菌遗传操作方法的不足,为这一重要工业微生物的多样化细胞工厂的构建提供了极大便利。此外,由于噬菌体位点特异性重组系统广泛分布且具有一定的普适性,该方法也可拓展到其他梭菌或固碳微生物的改造中,从而有效加速一碳气体生物转化利用的应用研究。
黄鹤副研究员为本论文的第一作者,顾阳研究员为共同通讯作者。该研究获得了国家自然科学基金、上海市科委“科技创新行动计划”基础研究领域项目、中科院青年创新促进会等资助。
论文链接:https://doi.org/10.1016/j.ymben.2019.01.005
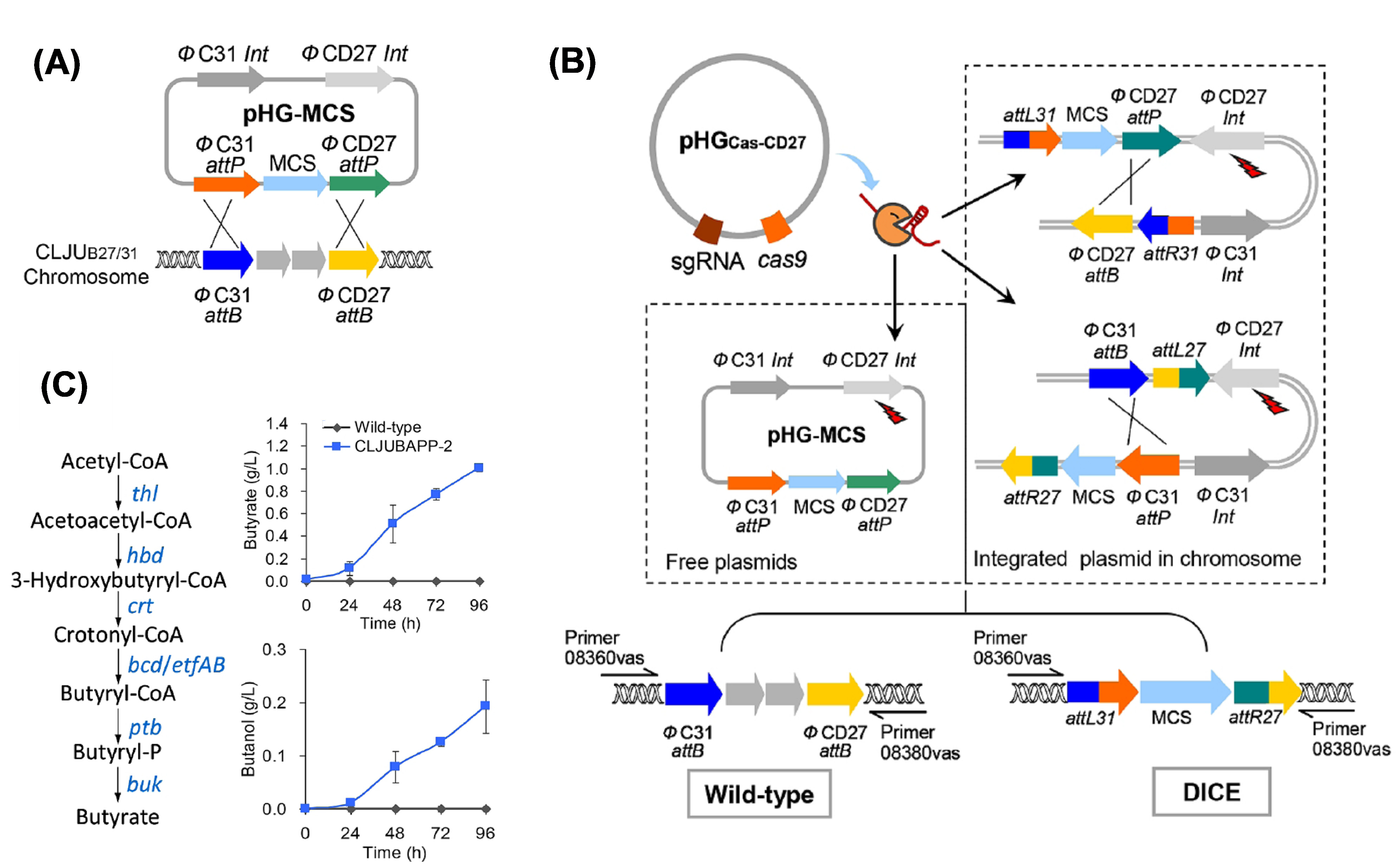
图1 基于噬菌体特异性整合系统的食气梭菌外源DNA染色体插入技术