姜卫红研究组建立基于细胞内源性群体感应和CRISPRi的新型多靶点动态调控系统
2020年 7月16日,国际学术期刊Nucleic Acids Research在线发表了中国科学院分子植物科学卓越创新中心姜卫红研究组题为“Developing an endogenous quorum-sensing based CRISPRi circuit for autonomous and tunable dynamic regulation of multiple targets in Streptomyces”的研究论文。该研究在链霉菌中创建了一种便捷、普适、自发驱动的动态调控系统,它可以同时对多个基因进行强度可调的动态转录干预,有效地提高了目的产物的合成效率,为微生物天然产物细胞工厂的研究与改造提供了一种新策略。
随着合成生物学的快速发展,传统的基因敲除和过表达等静态代谢工程手段的局限性逐渐显现,对新技术、新策略的需求日益迫切。能够时空特异性响应细胞环境变化的动态调控系统应运而生,已成为优化微生物产品效价的有用工具。然而,现有的动态调控系统往往需要通过繁琐的测试和优化过程,也很难同时对多个关键节点进行差异化控制,这阻碍了它们在工业微生物中的推广应用。链霉菌作为一类重要的工业微生物,能够产生复杂多样的活性天然产物,如目前广泛应用的抗生素、抗肿瘤药物和免疫抑制剂等,为人类健康、农业和畜牧业的发展作出了巨大贡献,但目前该菌的遗传操作主要通过静态代谢工程,有局限性。
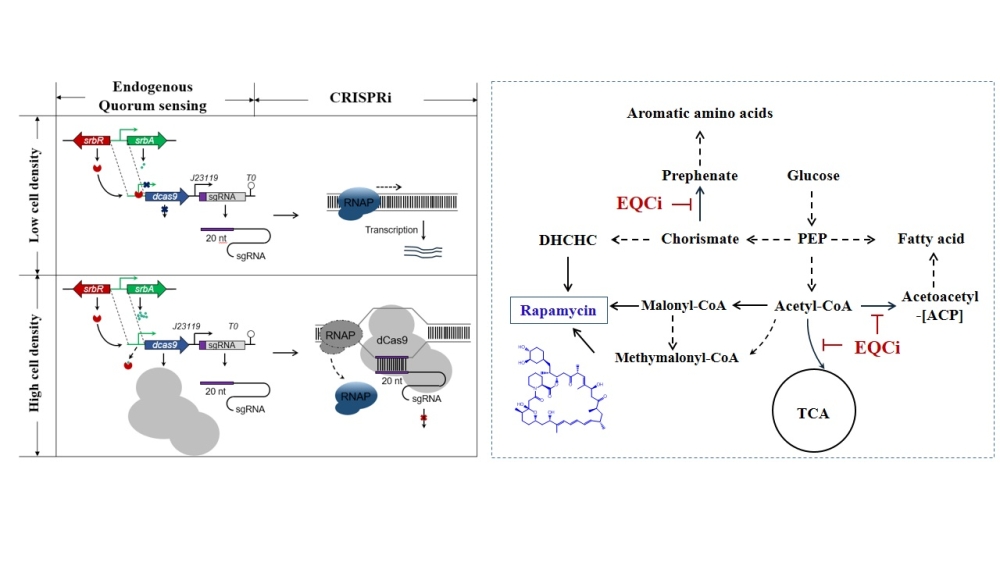
在该研究中,作者在链霉菌中设计构建了一种新型的动态调控系统EQCi,通过使用γ-丁内酯信号分子响应的启动子驱动dCas9基因的表达,巧妙地将内源性群体感应系统与基因转录抑制技术(CRISPRi)进行有机偶联。EQCi整合了二者的技术优点,能够同时对代谢途径中多个基因进行全自动、精细动态调节,并且操作简单、适用范围广。作者选择具有抗衰老、抗癌和免疫抑制等生物活性的重要天然化合物—雷帕霉素作为目标产物,利用EQCi重塑了雷帕霉素工业菌株—雷帕链霉菌的初级代谢网络。首先,通过分别下调三羧酸循环途径、脂肪酸合成途径和莽草酸途径三个初级代谢途径中关键节点基因的转录,以增加雷帕霉素生物合成的前体供应,显著提高了其效价。进一步使用EQCi系统对三个途径的代谢流进行组合干预,并对每个节点的控制强度进行精细微调,获得的最佳工程菌株中雷帕霉素效价达到1836±191 mg/l,为目前报道的最高水平。与出发菌株相比,提高了约660%。此外,作者还在另一株链霉菌—天蓝色链霉菌中测试了EQCi系统,通过动态控制三羧酸循环中的关键节点,亦大幅提高了次级代谢产物放线紫红素的产量。这些研究结果说明了该动态调控策略的有效性。
对比实验表明,静态调控介导的初级代谢抑制策略会导致细胞生长受阻、雷帕霉素产量急剧下降,而基于EQCi系统的动态新策略可以在不影响细胞生长的情况下显著提高雷帕霉素的效价,可见该系统有效地平衡了初级代谢和产物合成(次级代谢)的代谢流分配。EQCi系统为链霉菌来源的重要次级代谢天然产物细胞工厂的构建提供了一种高效、通用的优化策略,并有望拓展到其他工业微生物的分子育种与改造。
博士研究生田进忠为该论文的第一作者,姜卫红研究员和上海师范大学芦银华研究员为共同通讯作者。该研究得到了科技部、国家自然科学基金委、中国科学院等相关经费资助。此项工作还得到浙江医药股份有限公司的大力支持。
论文链接:https://academic.oup.com/nar/article/doi/10.1093/nar/gkaa602/5872467?searchresult=1