王二涛研究组与合作者提出“农业精准微生物组”概念
2022年10月7日,国际知名学术期刊Nature Communications在线发表了中国科学院分子植物科学卓越创新中心王二涛研究组和深圳华大生命科学院合作的题为“GWAS, MWAS and mGWAS provide insights into precision agriculture based on genotype-dependent microbial effects in foxtail millet”的研究论文。该研究以谷子为模型揭示了基因型、根系微生物组与农艺性状之间的互作网络,提出植物“农业精准微生物组”概念,为农业微生物组的精准施用奠定了理论基础。
全基因组关联研究(genome-wide association studies, GWAS)极大地促进了我们对植物基因型-表型关联的理解,但基因型往往不能完全解释种群内的表型变异。植物根系微生物群可以影响植物的表型及其对环境的适应性,被誉为植物的第二基因组,其对植物的表型和环境适应性的影响在遗传育种中被长期忽视。该研究基于827份谷子的基因型、根系生物群和生长及产量等表型数据,通过整合GWAS、微生物组关联研究(microbiome-wide association studies, MWAS)和微生物组-植物基因组关联研究(microbiome-genome association studies, mGWAS)等综合分析方法,探究了哪些“标记微生物”和哪些“标记基因”决定植物表型,对提高作物产量至关重要。
研究团队首先通过构建数学模型,量化了谷子基因型SNPs与根系微生物群对谷子生长和产量的影响。发现谷子基因型可以解释产量性状(如单株粒重)34.10%的变化,微生物可以解释43.31%的变化,基因型和微生物一起可以解释55.87%的变化,表明谷子基因型与微生物组共同决定谷子的生长和产量性状。
研究团队基于SNPs全基因组关联分析的方法,计算了不同微生物单元(operational taxonomic unit,OTU)的遗传力,发现根系微生物群的组成变化主要由植物免疫、代谢产物合成、激素信号传导以及养分吸收相关的基因来驱动。其中,宿主免疫基因FLS2和转录因子bHLH35与谷子根系微生物类群广泛关联。 通过比较分析发现谷子的遗传类群与高粱和玉米的遗传类群高度重叠,表明植物及其微生物群在宿主间具有相似的共进化模式。引人注目的是,研究团队通过构建“谷子基因型-标记微生物群-谷子农艺表型”的三角互作网络,并通过对网络的关键节点进行菌株接种验证,发现根系微生物调控作物农艺性状依赖宿主植物的基因型。基于此,研究团队提出了“农业精准微生物组”概念。
在未来的农业系统中,通过精准的微生物组管理设计高产的栽培品种和高效的微生物接种剂,向着改善植物-根系微生态互作的方向育种,将更加高效地促进农作物产量的提升。
深圳华大生命科学研究院王亚玉博士和中科院分子植物科学卓越创新中心博士后王孝林为该论文共同第一作者,深圳华大生命科学研究院刘欢研究员和中科院分子植物科学卓越创新中心王二涛研究员为论文共同通讯作者。研究获得国家自然科学基金委等项目的支持。
论文链接:https://www.nature.com/articles/s41467-022-33238-4
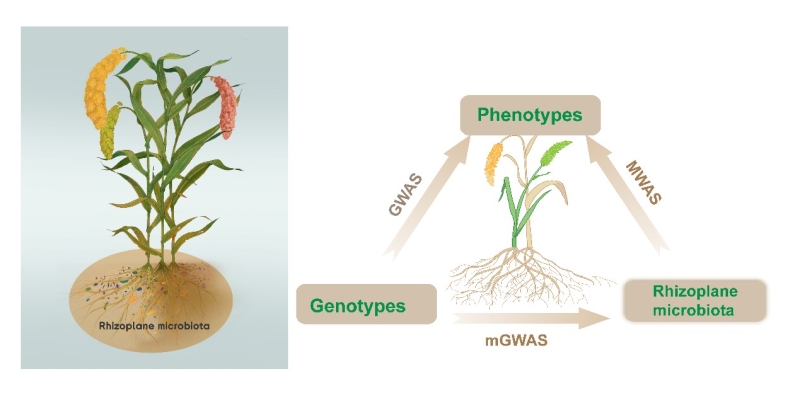
图. 谷子基因型-标记微生物-谷子农艺性状之间的互作网络